
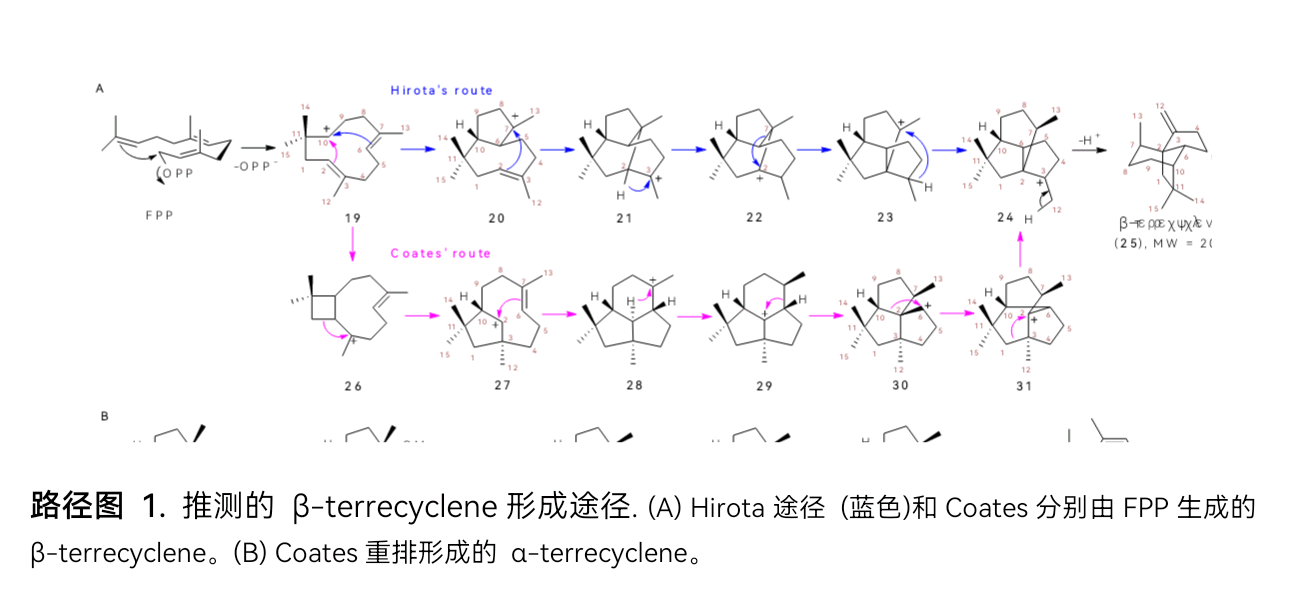
由于quadrane骨架的特殊结构与良好的生物学活性,其制备方法的研究受到广泛关注,其中合成化学家先后开发出20余条合成路线。然而由于该类化合物特殊的结构和成环方式,使得其化学合成步骤多,产率较低,在一定程度阻碍了先导药物的开发。通过合成生物学方法大量制备quadrane类天然产物,是解决这一问题的有效策略。同位素示踪实验表明,quadrane骨架的生物合成可能经历了两种完全不同的途径,即Hirota途径和Coates途径。尽管计算化学研究表明后者的可能性更高,但quadrane骨架的生物合成基因与其形成的确切酶学机制仍有待证实(路径图1)。
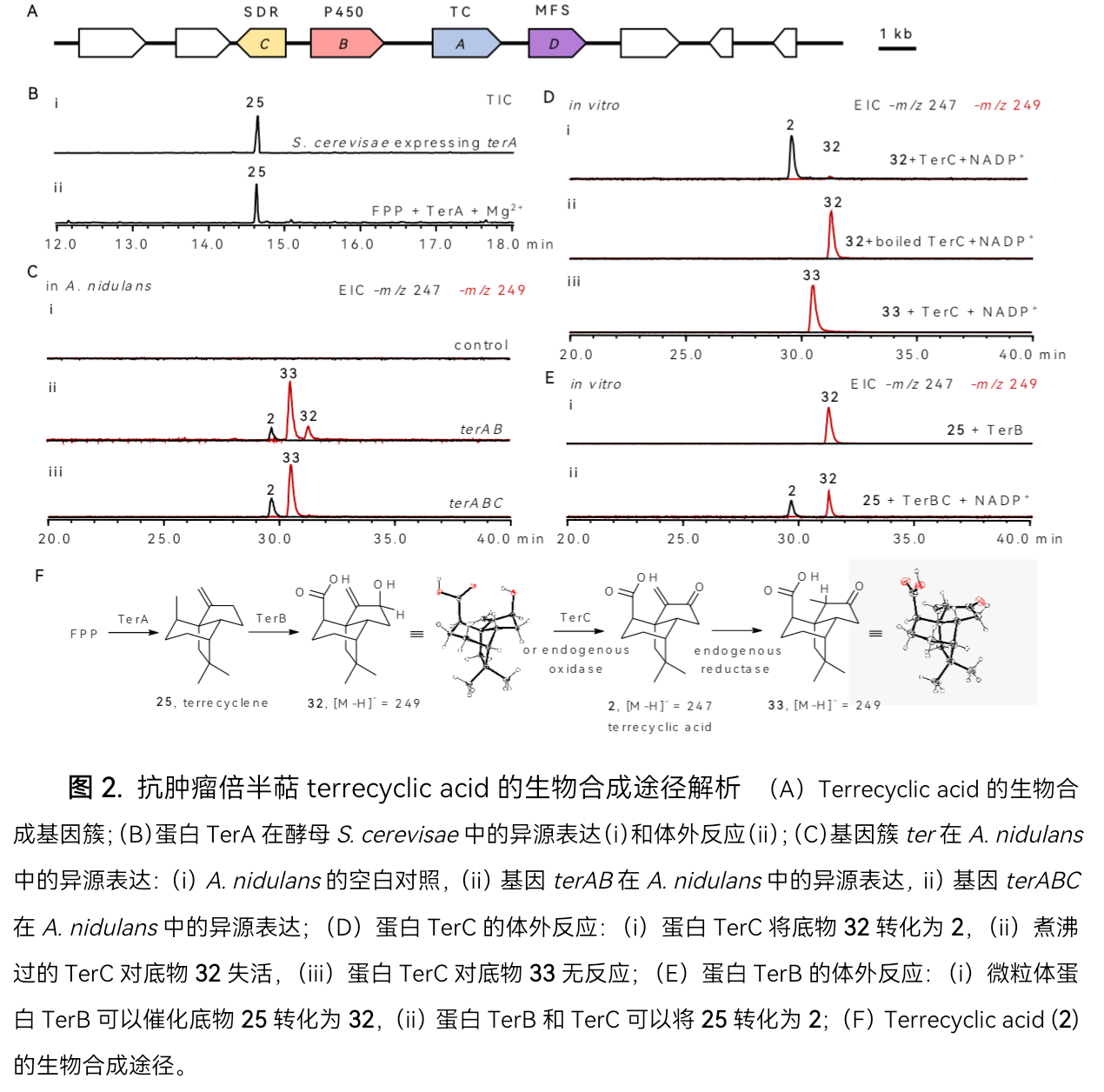
闫岩研究员团队通过对真菌基因组的挖掘,发现了该类化合物的生物合成基因簇ter,包括一个TC即terA、一个P450氧化基因(terB)、一个短链脱氢基因(terC)和一个主要协同转运因子超家族基因(terD),如图2A。通过异源表达、体内喂养、体外酶反应等研究,验证了基因簇中各基因的功能(图2B-2E),证实了其生物合成途径:即在环化酶TerA的作用下催化FPP产生化合物25,进而产物25被P450酶TerB羟化和羧基化产生化合物32,最后32被脱氢酶TerC催化形成终产物2,如图2F。
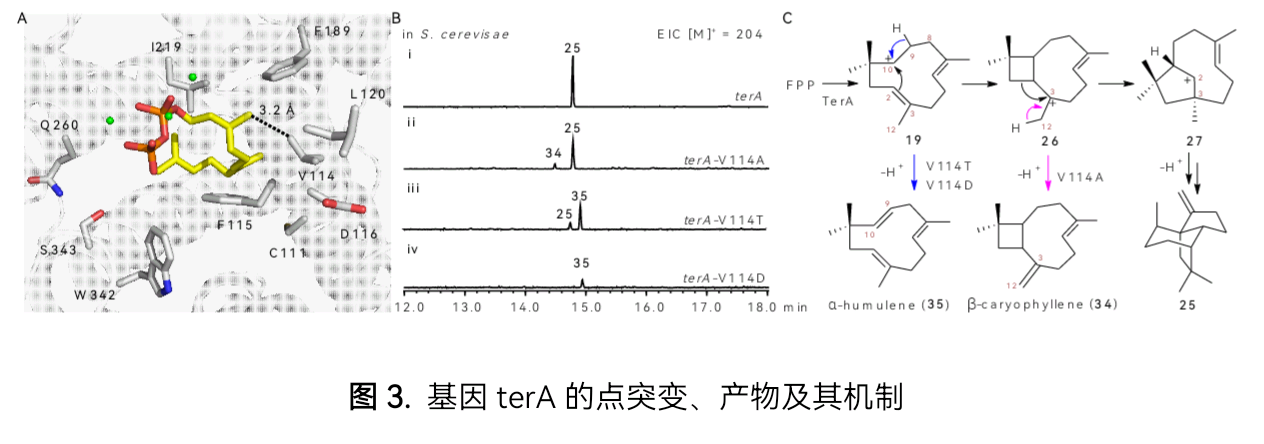
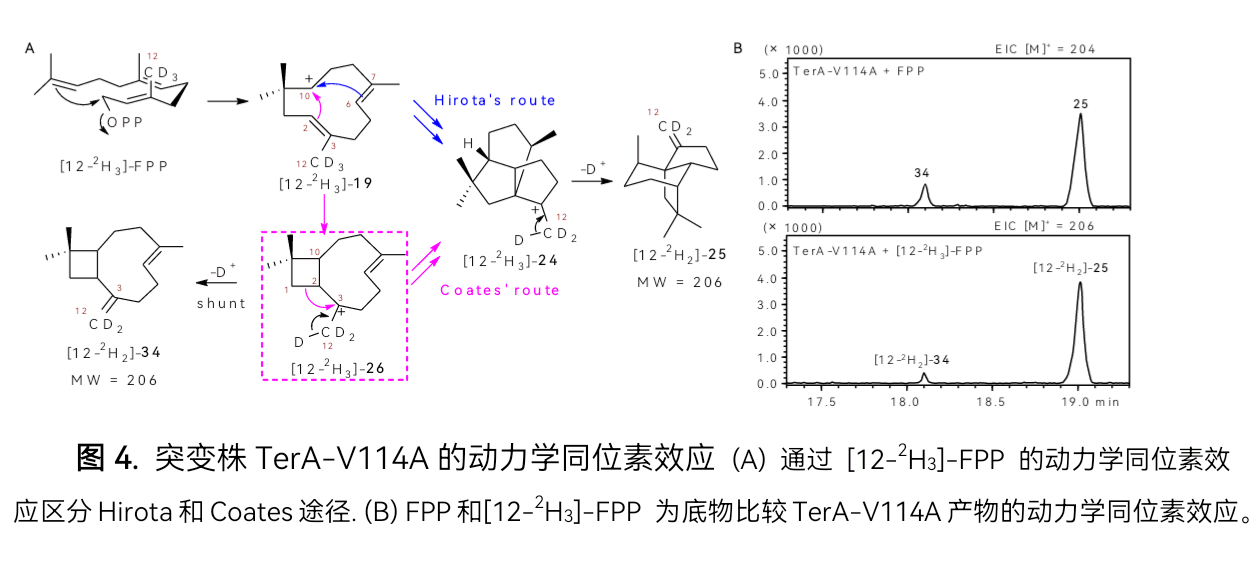
在发现新型倍半萜环化酶(β-terrecyclene合酶,TerA)可以催化法尼基焦磷酸(FPP)转化为β-terrecyclene(25)的基础上,通过对TerA的系列定点突变研究(图3),结合动力学同位素效应分析(图4),确证了环化过程为Coates途径,而非最早提出的Hirota途径。
通过以上研究,作者发现了抗菌、抗肿瘤quadrane骨架倍半萜terrecyclic acid的生物合成基因簇,以此为基础,解析了其通过β-terrecyclene环化酶TerA形成quadrane骨架,继而氧化、脱氢的后修饰的生物合成过程。Quadrane骨架的形成机制也通过对蛋白TerA的点突变和动力学同位素效应得以证实,从而解决了几十年来关于该类天然产物生物合成研究的谜团。该研究也为进一步以萜类环化酶TerA为探针,从真菌数据库中对quadrane类天然产物的发掘奠定了基础。